前言
说到测量程序的运行时间这件事,我最早的做法是在桌上摆个手机,打开秒表应用,右手在命令行里敲回车的同时左手启动秒表,看屏幕上程序跑完后再马上按停秒表,最后在纸上记下时间。后来我在 Linux 上学会了在命令开头添加一个 time,终于摆脱了手动计时的原始操作。这次就想总结一下迄今为止我用过的那些测量时间的工具/代码。
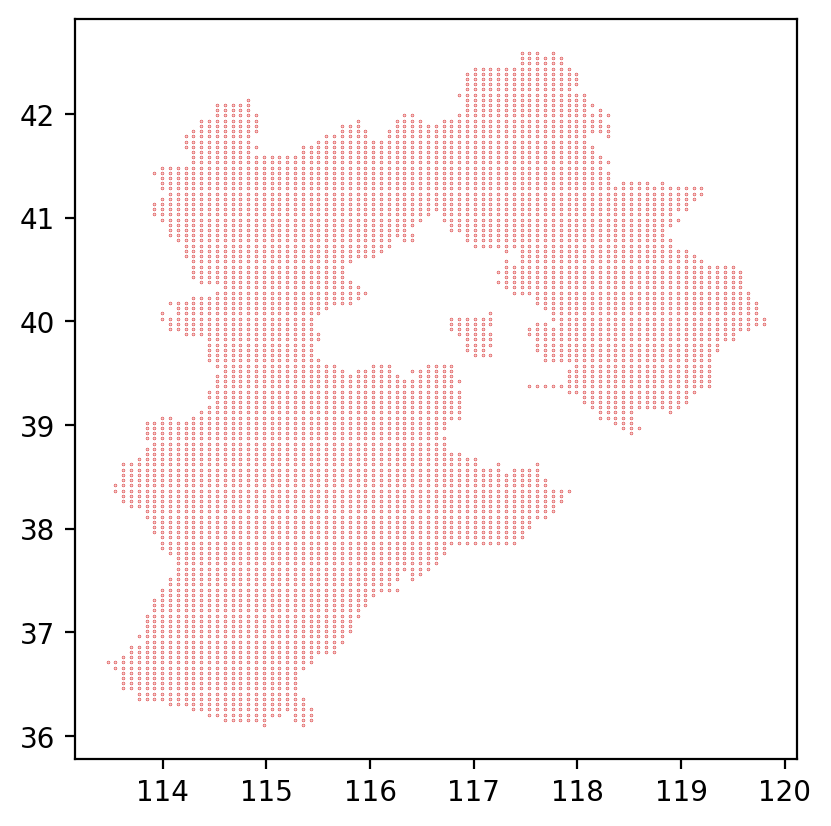
测试代码是读取河北省省界的 GeoJSON 文件,利用射线法判断网格点有没有落入省界内部,最后通过 Matplotlib 画出示意图并保存。GeoJSON 数据来自阿里云的 DataV.GeoAtlas。代码 test.py 的内容如下
import json
import numpy as np
import matplotlib.pyplot as plt
def contain(polygon, x, y):
'''判断点是否落入多边形中.'''
if polygon['type'] == 'Polygon':
coords_polygons = [polygon['coordinates']]
elif polygon['type'] == 'MultiPolygon':
coords_polygons = polygon['coordinates']
else:
raise ValueError('输入不是多边形')
# 对每个多边形应用射线法.
for coords_polygon in coords_polygons:
flag = False
for coords_ring in coords_polygon:
for i in range(len(coords_ring) - 1):
x0, y0 = coords_ring[i]
x1, y1 = coords_ring[i + 1]
if y0 < y <= y1 or y1 < y <= y0:
if x < (x1 - x0) / (y1 - y0) * (y - y0) + x0:
flag = not flag
if flag:
return flag
return False
def main():
with open('河北省.json', encoding='utf-8') as f:
geoj = json.load(f)
hebei = geoj['features'][0]['geometry']
xs = []
ys = []
for x in np.linspace(110, 125, 200):
for y in np.linspace(35, 45, 200):
if contain(hebei, x, y):
xs.append(x)
ys.append(y)
fig, ax = plt.subplots()
ax.set_aspect('equal')
ax.plot(xs, ys, 'o', c='C3', ms=0.2)
fig.savefig('hebei.png', dpi=200, bbox_inches='tight')
plt.close(fig)
if __name__ == '__main__':
main()
输出图片为
系统命令
Linux 的 time
在 Linux 的 Bash 中通过在命令前加上 time,即可在命令执行结束后打印出三种耗时
(base) laptop@zhajiman:/code$ time python test.py
real 0m22.993s
user 0m19.905s
sys 0m0.096s
其中 real 指总耗时(墙上时钟经过的时间,即 wall time),user 指用户态代码耗费的 CPU 时间,sys 指系统态代码耗费的 CPU 时间。一般看 real 的数值即可,关于三种时间的解释可见 linux time命令详解与坑。
Windows 的 Measure-Command
Windows 中类似的命令是 Powershell 的 Measure-Command 命令
(base) PS D:\code> Measure-Command {python test.py}
Days : 0
Hours : 0
Minutes : 0
Seconds : 16
Milliseconds : 442
Ticks : 164424518
TotalDays : 0.000190306155092593
TotalHours : 0.00456734772222222
TotalMinutes : 0.274040863333333
TotalSeconds : 16.4424518
TotalMilliseconds : 16442.4518
打印出了各种单位的耗时,一般看其中的 TotalSeconds 即可。不过该命令会吞掉 Python 程序本身的打印结果,这时可以通过管道在测量结束后把内容再打印出来
Measure-Command {python test.py | Out-Default}
显然 Linux 的 time 命令用起来要方便的多……
IPython 的魔法命令
%run
IPython 中的 %run 命令可以直接执行脚本文件,加上 -t 参数时会输出耗时
In [1]: %run -t test.py
IPython CPU timings (estimated):
User : 16.17 s.
System : 0.00 s.
Wall time: 16.37 s.
输出结果跟 Linux 的 time 命令很像,不过文档说 Windows 下的 System 时间直接设成了 0。加上 -N <N> 参数可以重复执行 <N> 次,输出里会多出总时间和平均时间之分。
%time
%time 会打印出单条语句或表达式的耗时
In [2]: %time main()
CPU times: total: 16.6 s
Wall time: 16.8 s
结果由 CPU 时间和墙上时间组成。
%timeit
类似 %time,但为了得到精准的测量结果,会自动测量多次,以得到较为准确的平均耗时,还会为打印出来的结果挑选合适的单位(秒、毫秒、微秒等)。例如
In [3]: with open('河北省.json', encoding='utf-8') as f:
...: geoj = json.load(f)
...: hebei = geoj['features'][0]['geometry']
...:
In [4]: %timeit contain(hebei, 115, 40)
284 µs ± 4.63 µs per loop (mean ± std. dev. of 7 runs, 1,000 loops each)
%timeit 默认跑 7 组,每组自动设置成了 1000 次循环,最后计算平均耗时和标准差。加上 -n <N> 可以指定循环 <N> 次,-r <R> 可以指定跑 <R> 组。例如测一次 main 函数的耗时
In [5]: %timeit -n 1 -r 1 main()
15.4 s ± 0 ns per loop (mean ± std. dev. of 1 run, 1 loop each)
无论是 %time 还是 %timeit 都有一个缺陷:只能在 IPython 顶层的作用域中使用。例如我想测试 contain(hebei, x, y) 对于单点的速度,因为变量 hebei 在函数 main 的作用域里,而外部没有,所以我在使用 %timeit 前又在全局新建了 hebei 变量。我还试过设置断点通过 ipdb 跳到函数内,结果发现此时 %timeit 用不了,不知道读者有没有比较好的解决方法。
标准库的 time 模块
time 和 perf_counter 函数
time 模块的一个主要功能就是获取当前时间,所以只要在想计时的代码块开头获取一次时间,再在结尾获取一次,最后计算二者的差值就能得到代码块的耗时。例如
import time
if __name__ == '__main__':
t0 = time.time()
main()
t1 = time.time()
dt = t1 - t0
print(f'{dt:.1f}', 's')
打印结果为 15.4 s。其中 time.time 函数没有参数,在 Windows 和 Linux 平台上调用后返回 Unix 时间戳(自 1970-01-01 00:00:00 UTC 以来的秒数)。这个函数的问题是与系统时间相关联,如果两次调用之间系统时间发生了变动(例如在任务栏里手动修改、联网自动校正等),那么第二次调用的结果也会跟着变化,最后计算出错误的 dt。另外该函数在 Windows 平台的时间分辨率也比较低,会测不准耗时较短的语句。
time.perf_counter 是 Python 3.3 起引入的新函数,名字是 performance counter 之意,即专门用来测量性能(耗时)的计时器。调用后返回的浮点数本身没有明确的意义,只有两次调用时结果的差值有意义,表示过程中经过的秒数。相比 time,perf_counter 的时间分辨率更高,且不受系统时间变动的影响,结果始终保证单调递增。所以在测量程序耗时中更推荐使用 perf_counter。
if __name__ == '__main__':
t0 = time.perf_counter()
main()
t1 = time.perf_counter()
dt = t1 - t0
print(f'{dt:.1f}', 's')
另外 time 模块里还有一个 process_time 函数,说是能测量当前进程的用户态和系统态 CPU 时间之和,且不包含睡眠时间(例如调用 time.sleep 函数)。但我测试后发现测出来的时间经常比 perf_counter 的结果要大,实在让人摸不着头脑,所以这里就不多介绍了。
装饰器
直接在程序中插入 perf_counter 的用法虽然很灵活,但改起来十分麻烦。如果只想测量函数的耗时,使用装饰器语法更方便:无需修改函数体或主程序,只需在定义函数的 def 语句前插入一行,就能为函数增加计时功能。下面编写一个带可选参数的计时装饰器
import time
import functools
def timer(func=None, *, prompt=True, fmt=None, prec=None, out=None):
'''
计时用的装饰器.
Parameters
----------
func : callable
需要被计时的函数或方法.
prompt : bool
是否打印计时结果.
fmt : str
打印格式. %n表示函数名, %t表示耗时.
prec : int
打印时的小数位数.
out : list
收集耗时的列表.
'''
if fmt is None:
fmt = '[%n] %t s'
if func is None:
return functools.partial(
timer, prompt=prompt, fmt=fmt, prec=prec, out=out
)
@functools.wraps(func)
def wrapper(*args, **kwargs):
t0 = time.perf_counter()
result = func(*args, **kwargs)
t1 = time.perf_counter()
dt = t1 - t0
# 是否打印结果.
if prompt:
st = str(dt) if prec is None else f'{dt:.{prec}f}'
info = fmt.replace('%n', func.__name__).replace('%t', st)
print(info)
# 是否输出到列表中.
if out is not None:
out.append(dt)
return result
return wrapper
装饰器相关的知识可见 入门Python装饰器。用法是在 main 函数前一行加上 @timer 即可,还可以通过参数控制装饰器的输出
results = []
@timer(fmt='Time spent by %n is %t s', prec=1, out=results)
def main():
<函数体省略>
输出为
Time spent by main is 16.9 s
同时耗时被保存到了全局的 results 列表中。如果想要测量的函数并非定义在当前文件里(例如第三方包里的函数),那么可以通过包装目标函数来实现。例如测量 figsave 方法的用时
timer(prec=1)(fig.savefig)('hebei.png', dpi=200, bbox_inches='tight')
输出为
[savefig] 0.148 s
标准库的 cProfile 模块
time 模块适合测量代码块或函数的总耗时,如果想深入了解组成代码的所有函数各占了多长时间,推荐使用标准库的 cProfile 模块。该模块能够对程序进行性能剖析(profile),详尽给出各种函数和方法被调用的次数和耗时。但也因为监控的层级过于深入,会在一定程度上拖慢原程序的运行速度。所以该模块适合分析程序内各部分的相对用时,而不适合做精确的性能比较(benchmark)。
命令行调用
直接在命令行调用会对整个脚本进行测量,无需对代码进行任何修改
python -m cProfile [-o output_file] [-s sort_order] myscript.py
-o 表示将剖析结果输出为二进制文件,如果不给出就将结果以表格的形式打印在屏幕上。-s 表示如果不输出文件,那么可以按 pstats 模块 Stats.sort_stats 方法的规则对打印的表格记录进行排序。由于表格一般巨长无比,所以打印到屏幕上意义不大,我会用管道输出到文本文件后再用编辑器来看。例如在 PowerShell 中可以
python -m cProfile -s cumtime test.py | Out-File -FilePath test.txt
表格前 20 行长这个样子
3372760 function calls (3338684 primitive calls) in 19.628 seconds
Ordered by: cumulative time
ncalls tottime percall cumtime percall filename:lineno(function)
782/1 0.003 0.000 19.629 19.629 {built-in method builtins.exec}
1 0.000 0.000 19.629 19.629 test.py:1(<module>)
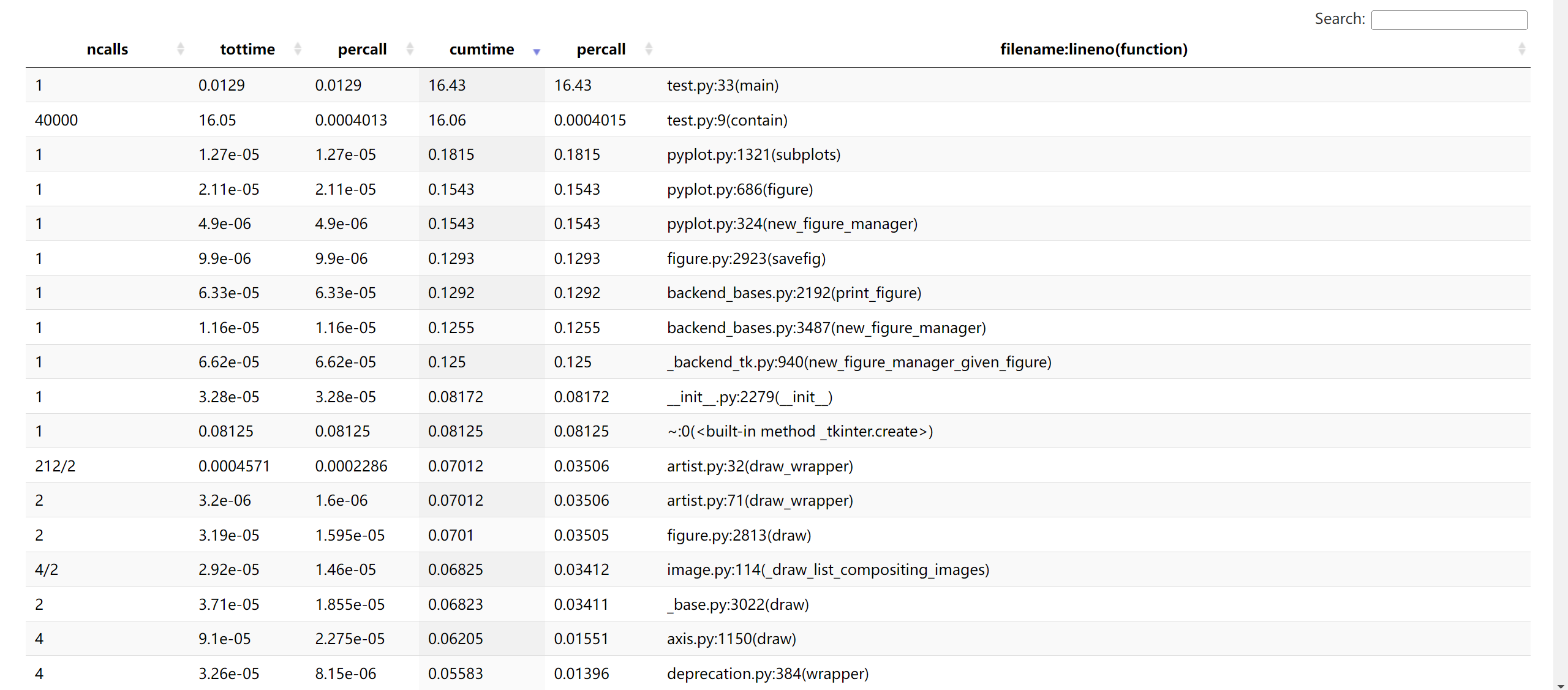
1 0.028 0.028 17.907 17.907 test.py:33(main)
40000 17.519 0.000 17.532 0.000 test.py:9(contain)
96 0.003 0.000 3.383 0.035 __init__.py:1(<module>)
871/15 0.006 0.000 1.760 0.117 <frozen importlib._bootstrap>:1022(_find_and_load)
868/15 0.004 0.000 1.760 0.117 <frozen importlib._bootstrap>:987(_find_and_load_unlocked)
833/16 0.004 0.000 1.755 0.110 <frozen importlib._bootstrap>:664(_load_unlocked)
773/15 0.002 0.000 1.754 0.117 <frozen importlib._bootstrap_external>:877(exec_module)
1106/16 0.001 0.000 1.754 0.110 <frozen importlib._bootstrap>:233(_call_with_frames_removed)
535/25 0.001 0.000 1.659 0.066 {built-in method builtins.__import__}
1086/417 0.002 0.000 1.264 0.003 <frozen importlib._bootstrap>:1053(_handle_fromlist)
2652/2432 0.038 0.000 0.926 0.000 {built-in method builtins.__build_class__}
1 0.000 0.000 0.883 0.883 pyplot.py:1(<module>)
101 0.001 0.000 0.690 0.007 artist.py:128(_update_set_signature_and_docstring)
可以看到加了 cProfile 后程序耗时从 16 秒上升至 19 秒,期间共发生 3372760 次函数调用(不算递归的原始调用是 3338684 次)。表格各列的意义如下:
ncalls:函数调用的次数。正斜杠区分总次数和原始调用。tottime:每次调用的耗时之和,但如果函数内调用了其它函数,刨除掉在子函数中经过的时间。percall:tottime除以ncalls的值。cumtime:每次调用的耗时之和,包含花费在子函数上的时间。percall:cumtime除以原始调用次数的值。filename:lineno(function):函数所在的文件名、定义所在的行号,和函数名。
-s 选项可以按这些列名进行排序,文件名和行号的话用 filename 和 line。表格中总耗时排第一的是内置方法 exec,可能是 cProfile 需要用 exec 执行 test.py 中的语句;第二是作为整个模块的 test.py,再是 main 函数;第四的 contain 函数调用 200 * 200 = 40000 次,耗时 17 秒,说明整个程序中最耗时的操作就是判断点是否落入多边形。而画图相关的函数在表格中的排位并不算高。
程序内调用
如果想要指定测量范围,就需要在程序中显式调用 cProfile 模块。测量单条语句的耗时可以用:
cProfile.run(command, filename=None, sort=-1):command是用字符串表示的 Python 语句,该方法会用exec函数执行该语句并测量其耗时。filename参数用于将结果输出为文件,缺省时会打印出结果表格。sort参数类似上一节,对打印结果进行排序。cProfile.runctx(command, globals, locals, filename=None, sort=-1):传入run的语句仅能引用全局作用域中的对象,而runctx可以指定全局作用域和局部作用域的字典。
例如测量 main() 这一句的耗时,输出与上一节类似
import cProfile
if __name__ == '__main__':
cProfile.run('main()', sort='cumtime')
测量代码块的耗时需要构造 cProfile.Profile 对象,它相当于一个计时器,通过调用方法来开关,会把计时结果保存下来,之后可以选择打印或输出文件。具体方法为:
enable():开始测量。disable():结束测量。print_stats(sort=-1):在内部创建一个Stats对象并用它打印表格,用sort参数进行排序。dump_stats(filename):将测量结果输出到文件。run(cmd):类似cProfile.run,但没了打印和输出文件功能。runctx(cmd, globals, locals):类似cProfile.runctx。runcall(func, /, *args, **kwargs):相当于先enable(),再跑func(*args, **kwargs),然后disable()。
以画图部分的代码块为例,先在开头开启计时器,再在结尾停止计时
profile = cProfile.Profile()
profile.enable()
fig, ax = plt.subplots()
ax.set_aspect('equal')
ax.plot(xs, ys, 'o', c='C3', ms=0.2)
fig.savefig('hebei.png', dpi=200, bbox_inches='tight')
plt.close(fig)
profile.disable()
profile.print_stats('cumtime')
另外 Profile 类支持上下文管理器,允许用 with 语句控制计时器的开关。所以上例可以改写为
with cProfile.Profile() as profile:
fig, ax = plt.subplots()
ax.set_aspect('equal')
ax.plot(xs, ys, 'o', c='C3', ms=0.2)
fig.savefig('hebei.png', dpi=200, bbox_inches='tight')
plt.close(fig)
profile.print_stats('cumtime')
前 20 行结果为,可以看到保存图片的语句耗时最长
199075 function calls (195323 primitive calls) in 0.454 seconds
Ordered by: cumulative time
ncalls tottime percall cumtime percall filename:lineno(function)
1 0.000 0.000 0.251 0.251 figure.py:2923(savefig)
1 0.000 0.000 0.251 0.251 backend_bases.py:2192(print_figure)
1 0.000 0.000 0.192 0.192 pyplot.py:1321(subplots)
1 0.000 0.000 0.165 0.165 pyplot.py:686(figure)
1 0.000 0.000 0.165 0.165 pyplot.py:324(new_figure_manager)
1 0.000 0.000 0.130 0.130 backend_bases.py:3487(new_figure_manager)
1 0.000 0.000 0.129 0.129 _backend_tk.py:940(new_figure_manager_given_figure)
2 0.000 0.000 0.125 0.063 artist.py:71(draw_wrapper)
212/2 0.001 0.000 0.125 0.063 artist.py:32(draw_wrapper)
2 0.000 0.000 0.125 0.063 figure.py:2813(draw)
4/2 0.000 0.000 0.122 0.061 image.py:114(_draw_list_compositing_images)
2 0.000 0.000 0.122 0.061 _base.py:3022(draw)
4 0.000 0.000 0.113 0.028 axis.py:1150(draw)
4 0.000 0.000 0.111 0.028 deprecation.py:384(wrapper)
2 0.000 0.000 0.109 0.054 backend_bases.py:1595(wrapper)
装饰器
编写一个测量函数用时并保存结果的装饰器
import cProfile
import functools
def cprofiler(filename):
'''cProfile的装饰器. 保存结果到指定路径.'''
def decorator(func):
@functools.wraps(func)
def wrapper(*args, **kwargs):
with cProfile.Profile() as profile:
result = func(*args, **kwargs)
profile.dump_stats(filename)
return result
return wrapper
return decorator
使用方法为
@cprofiler('main.prof')
def main():
<函数体省略>
可视化结果
cProfile 模块输出的二进制文件可以用 pstats 模块的 Stats 类进行读取,它相当于一个表格对象,能对记录进行复杂的排序、只打印前 n 条记录等。还可以直接用记录了测量结果的 Profile 对象构造 Stats 对象。Profile.print_stats 方法其实就是借助 Stats.print_stats 实现的。不过个人觉得这个模块挺鸡肋,不如直接 Profile.print_stats('cumtime') 结合管道导出文本文件,然后再在文本编辑器中查看。
除此之外,另一个直观查看结果的方式就是用第三方包做可视化。这里介绍 snakeviz 包,通过 pip 或 conda 即可安装,使用方法非常简单,在命令行执行
snakeviz main.prof
就能解析 main.prof 文件,在弹出的网页里展示可视化结果。下图是各函数耗时的冰柱图(icicle):最顶层的长条矩形是被 cProfile.Profile 包裹的 main 函数,下一层则将上一层的矩形细分为不同颜色的子矩形,对应于 main 的函数体中被调用的各种函数,矩形长度正比于耗时。再下一层又会细分上一层的函数调用,如此不断深入,直至某个函数仅由内置语句和函数组成,或调用了其它语言的库文件。最后图片会自上而下形成一个个小山丘,又似冬天屋檐上结成的冰柱,各函数的耗时情况一目了然。Snakeviz 的网页里可以用鼠标点击感兴趣的矩形,会以这个函数为顶层放大冰柱图的细节,之后可以点击左边的 Reset Zoom 按钮返回初始状态。
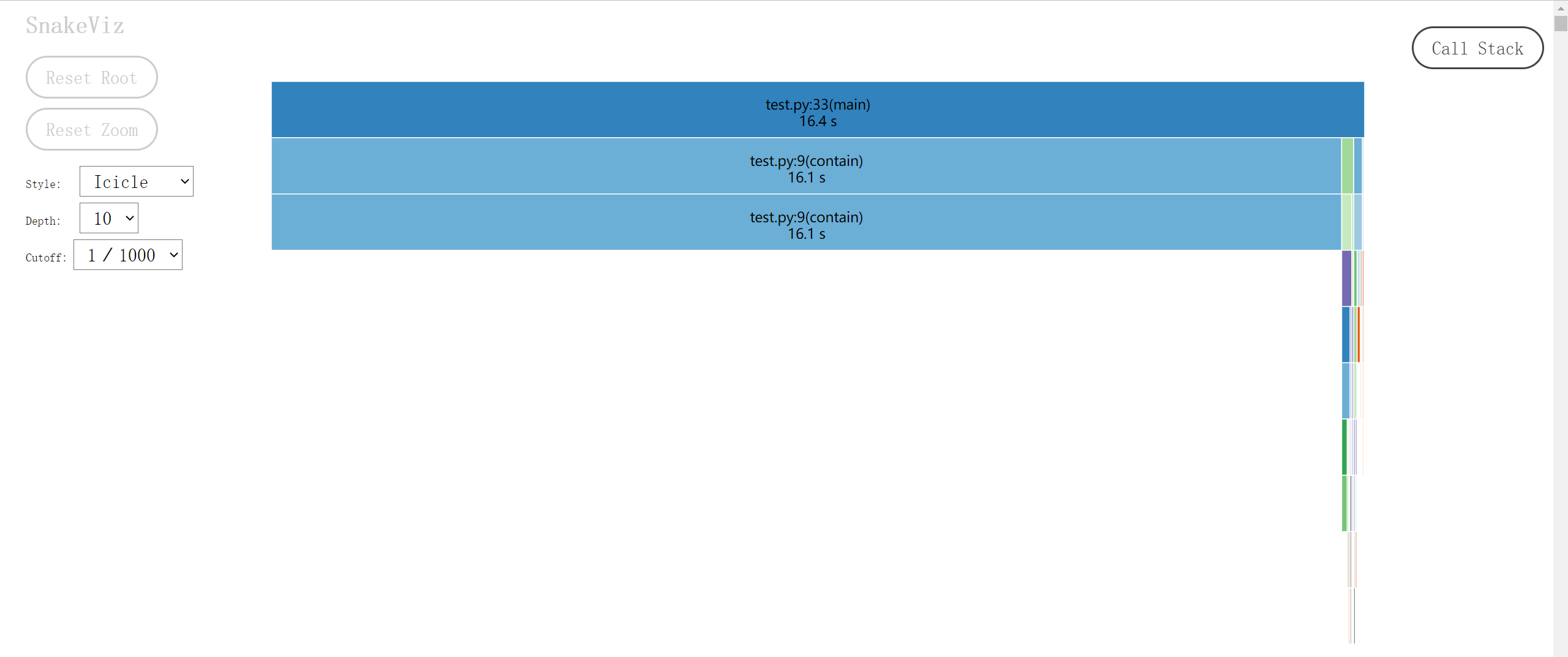
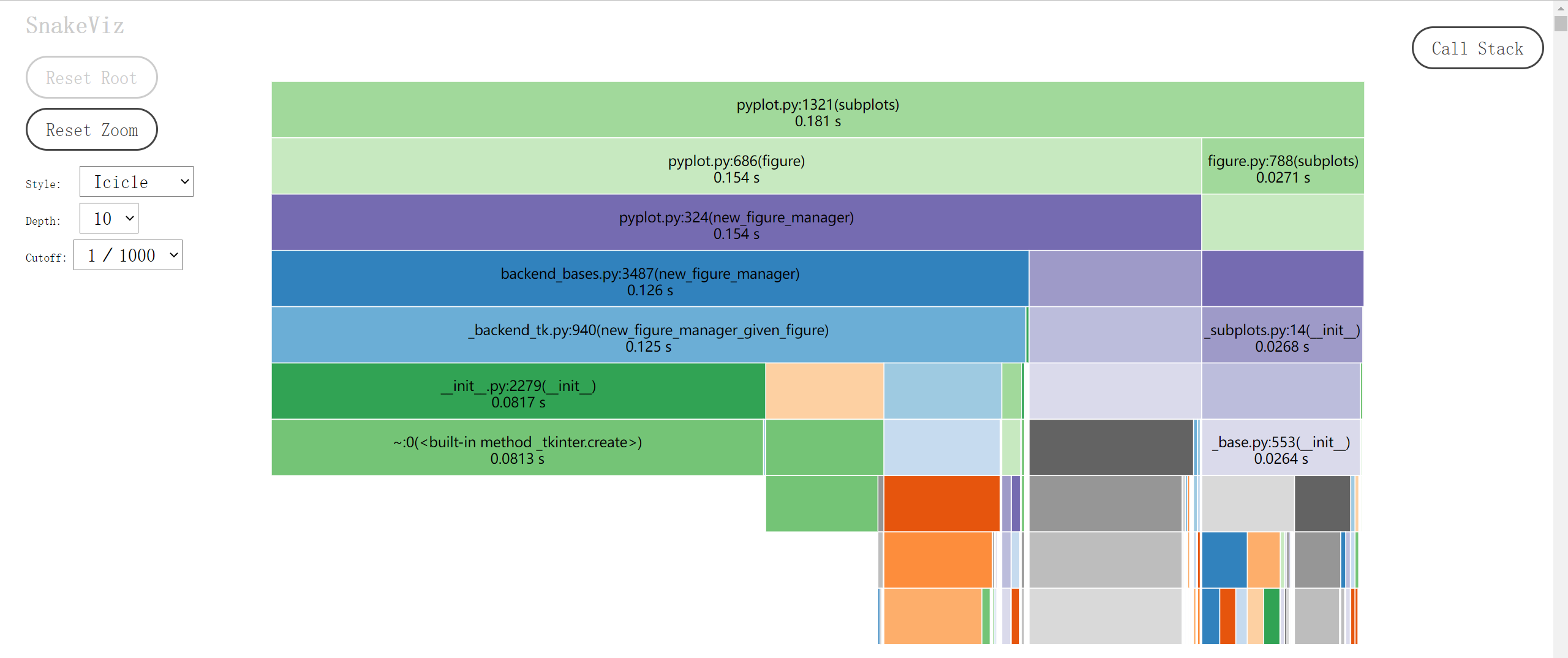
网页下半部分还有表格结果,点击列名以排序,点击行以做出该函数为顶层的冰柱图。这样一来连打印表格的 Profile.print_stats 都可以省去了。
除 snakeviz 外,还有 gprof2dot、flameprof 等工具可选。
line_profiler 模块
cProfile 只能监测函数和方法的耗时,并且会深入到最底层的调用,有时我们并不需要如此详细的信息,只是想了解每一行的耗时。这种情况下第三方模块 line_profiler 可能会更合适,正如它的名字所示,测量的是函数体每一行语句的耗时。同样是 pip 或 conda 安装。
程序内调用
line_profiler.LineProfiler 只提供对函数的测量功能,在构造时需要传入目标函数,也可以后面再添加
from line_profiler import LineProfiler
profile = LineProfiler(f, g)
profile.add_function(h)
常用的方法有:
run(cmd):测量一条语句的耗时。但如果语句中不含对目标函数的调用,那就啥也测不到。runctx(cmd, globals, locals):可以传入环境的run。runcall(func, *args, **kw): 测量func(*args, **kw)耗时。当然前提是之前添加过func函数。enable_by_count():据文档说是解决了嵌套安全性的enable。disable_by_count():同上。print_stats(self, stream=None, output_unit=None, stripzeros=False):打印结果,stream指定输出流(默认屏幕),output_unit指定结果中的时间单位(后面再解释),stripzeros指定是否隐藏未被调用的函数。dump_stats(self, filename):以二进制格式导出文件。
以 main 函数为例
if __name__ == '__main__':
profile = LineProfiler(main)
profile.enable_by_count()
main()
profile.disable_by_count()
profile.dump_stats('main.lprof')
生成的二进制文件通过下面的命令打印
python -m line_profiler main.lprof
Timer unit: 1e-06 s
Total time: 60.5731 s
File: D:\code\python\profiler\test.py
Function: main at line 34
Line # Hits Time Per Hit % Time Line Contents
==============================================================
34 def main():
35 2 87.8 43.9 0.0 with open('河北省.json', encoding='utf-8') as f:
36 1 895.7 895.7 0.0 geoj = json.load(f)
37 1 1.4 1.4 0.0 hebei = geoj['features'][0]['geometry']
38
39 1 0.7 0.7 0.0 xs = []
40 1 0.5 0.5 0.0 ys = []
41 201 295.5 1.5 0.0 for x in np.linspace(110, 125, 200):
42 40200 59299.5 1.5 0.1 for y in np.linspace(35, 45, 200):
43 40000 60157225.0 1503.9 99.3 if contain(hebei, x, y):
44 5226 9045.1 1.7 0.0 xs.append(x)
45 5226 3818.1 0.7 0.0 ys.append(y)
46
47 1 188088.4 188088.4 0.3 fig, ax = plt.subplots()
48 1 34.6 34.6 0.0 ax.set_aspect('equal')
49 1 1567.8 1567.8 0.0 ax.plot(xs, ys, 'o', c='C3', ms=0.2)
50 1 145367.5 145367.5 0.2 fig.savefig('hebei.png', dpi=200, bbox_inches='tight')
51 1 7339.0 7339.0 0.0 plt.close(fig)
Line:行号。Hits:这一行被“命中”(执行)了几次。Time:这一行的总耗时。乘上开头的时间单位Timer unit后才是真实数值。Per Hit:Time/Hits的值,表示每次执行的平均耗时。% Time:百分数形式的Time。Line Contents:这一行语句的内容。
因为表格内容非常清晰易懂,所以不需要做什么可视化,并且也不会像 cProfile 那样出现一堆见都没见过的函数。令人大跌眼镜的是,被测量的 main 函数耗时从 16 秒暴增至 60 秒。暗示循环语句太多时测量会严重拖慢程序,幸好我们更关心的是 % Time 列的内容,即哪一行占的时间最多。显然 contain 占据了 99.3 %,这提示我们 contain 函数应该是优化的首要对象。
上面的用法还可以改写为
if __name__ == '__main__':
profile = LineProfiler(main)
profile.runcall(main)
profile.print_stats()
然后用管道输出文本结果,这样就可以省去中间的 test.lprof 文件和 python -m 命令。
装饰器
LineProfiler 类的实例可以直接用作装饰器,会自动通过 add_function 方法添加目标函数。例如
profile = LineProfiler()
@profile
def contain(polygon, x, y):
<函数体省略>
if __name__ == '__main__':
main()
profile.print_stats()
另外也可以自己实现一个保存结果的装饰器
def lprofiler(filename):
'''line_profiler的装饰器. 保存结果到指定路径.'''
def decorator(func):
@functools.wraps(func)
def wrapper(*args, **kwargs):
profile = LineProfiler(func)
result = profile.runcall(func, *args, **kwargs)
profile.dump_stats(filename)
return result
return wrapper
return decorator
不过自己写的版本有个毛病,反复调用被装饰的函数会重写结果文件,导致无法测量累积用时。
kernprof
kernprof 是 line_profiler 附带的一个脚本,作用是简化调用 LineProfiler 的流程,自动输出结果文件。在命令行里用它代替 Python 命令执行脚本时,会先构造一个名为 profile 的 LineProfiler 对象,并将其注入脚本的作用域。这样一来脚本开头无需添加 import 语句就能直接引用 profile 变量,接着用它装饰目标函数即可。这也是 line_profiler 文档的推荐用法。例如在脚本中加上一行
@profile
def main():
<函数体省略>
也不需要写什么 profile.dump_stats(filename),直接在命令行执行
kernprof -l test.py
就会在当前目录自动生成 test.py.lprof 文件。kernprof 的详细用法可以用 -h 选项查看,下面列举几条常用的:
-l, --line-by-line:给出时使用 line_profiler,否则使用 cProfile。-b, --builtin:是否把profile注入脚本。-l时默认开启。-o OUTFILE, --outfile OUTFILE:将结果输出至OUTFILE文件。不给出时默认以脚本名.prof或脚本名.lprof为名保存到当前目录。-v, --view:是否顺便打印出结果。
其中 -l 选项提到了可以使用 cProfile,kernprof 对 cProfile.Profile 进行了包装,为其增加了装饰器功能,用法跟前面一样。以测量代码块为例
with profile:
<代码块省略>
不加 -l,记得加 -b
kernprof -b test.py
会在当前目录自动生成 test.py.prof 文件,接着可以交给 snakeviz 去可视化。
结语
由简到繁总结一下上述工具的使用场景:
- 测量脚本用时:系统命令或 IPython 的
%run。 - 测量函数用时:IPython 的
%time、%timeit。 - 测量代码块用时:time 模块。
- 测量函数每一行的用时:line_profiler 模块。
- 测量函数或代码块所有调用的用时:cProfile 模块。
因笔者对 Jupyter Notebook 不熟,所以没能介绍 %time 和 %timeit 的 cell 版本。另外也没能测试多进程下各工具的表现,专门提到这个是因为笔者在实际操作中经历过 line_profiler 和 kernprof 在多进程脚本中失效的现象。以后有机会再总结一下这些内容。
参考资料
Point-In-Polygon Algorithm — Determining Whether A Point Is Inside A Complex Polygon
Windows equivalent to UNIX “time” command
IPython: Built-in magic commands
Understanding time.perf_counter() and time.process_time()
关于python中time.perf_counter() 与 time.process_time()分析与疑问
Python Cookbook: 9.6 带可选参数的装饰器